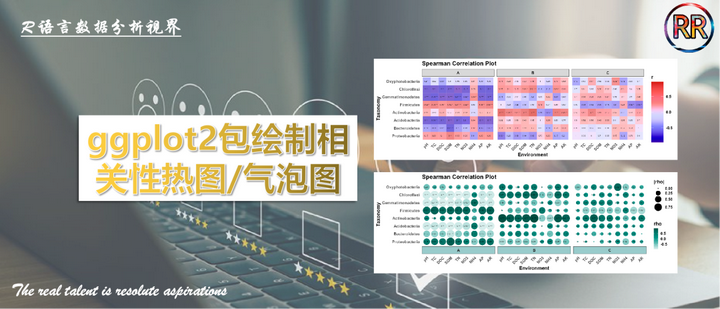
相关性分析热图:ggplot2包绘制环境因子和物种相关性热图/气泡图
上述代码中:我们载入了需要的R包,并读取了环境因子和物种数据。然后,我们使用psych包的corr.test函数执行了相关性分析,并将结果保存在r_result变量中。接下来,我们整理了相关性和p值的数据,并根据P值的不同范围,构建“***”并使用标记;在这段代码中,我们使用ggplot函数创建了一个基本的绘图对象,并使用geom_tile函数绘制了热图。大家好,今天我将介绍如何使用R语言进行环境
数据分析服务请访问以下链接:

数据和代码获取:请查看主页个人信息
大家好,今天我将介绍如何使用R语言进行环境因子和物种相关性分析,并使用ggplot2包绘制热图和气泡图,对相关性分析结果进行可视化展示。

相关性热图样式灵感来源于Microbiome杂志的一篇文章:


Step1:环境因子与物种数据相关性分析
rm(list=ls())
pacman::p_load(tidyverse,reshape2,psych)
# 载入环境/物种数据
env <- read.delim('env_table.txt', sep = '\t', row.names = 1)
taxonomy <- read.delim('spe_table.txt', sep = '\t', row.names = 1)
env <- env[rownames(taxonomy), ]
# 执行相关性分析
r_result <- corr.test(taxonomy, env, method = 'spearman')
r <-
r_result$r %>%
melt() %>%
set_names(c('tax', 'Index', 'r'))
p <-
r_result$p %>%
melt() %>%
set_names(c('tax', 'Index', 'P_value')) %>%
mutate(P_value_sig = case_when(P_value > 0.05 ~ " ",
P_value <= 0.05 & P_value > 0.01 ~ "*",
P_value <= 0.01 & P_value > 0.001 ~ "**",
P_value <= 0.001 ~ "***",
TRUE ~ NA_character_))
data <- cbind(r,p) %>% select(-(4:5))
head(data)
上述代码中:我们载入了需要的R包,并读取了环境因子和物种数据。然后,我们使用psych包的corr.test函数执行了相关性分析,并将结果保存在r_result变量中。接下来,我们整理了相关性和p值的数据,并根据P值的不同范围,构建“***”并使用标记;之后使用melt函数将数据进行重塑,以便后续的可视化处理。
Step2:所有样本整体相关性的热图简单展示
# 绘图主题
theme <- theme_bw(base_size = 7) +
theme(panel.grid.major = element_blank(),
text = element_text(face='bold'),
legend.key.size = unit(5, "pt"),
panel.grid.minor = element_blank(),
axis.ticks.y = element_blank(),
axis.text.x=element_text(angle=45,vjust=1, hjust=1))
ggplot(data,aes(x=tax, y=Index))+
geom_tile(aes(fill=r), color = 'white',alpha = 0.6) +
geom_text(aes(label = P_value_sig), color = 'black', size = 2) +
ggsci::scale_fill_gsea() +
theme(legend.position="top") +
theme +
xlab('') +
ylab('')
ggsave('heatmap_corr.png',height = 2, width = 2)
在这段代码中,我们使用ggplot函数创建了一个基本的绘图对象,并使用geom_tile函数绘制了热图。通过geom_text函数,我们还添加了对应的p值显著性标记。这张热图将展示环境因子和物种之间的相关性,并通过颜色填充来表示p值的显著性。
接下来,我们定义一个函数cal,用于计算不同组内物种与环境因子之间的相关性。代码如下:
Step3:定义分组相关性计算函数
# 定义一个函数,计算不同组内物种与环境因子之间的相关性
cal <- function(group){
# 提取分组样品名称
g <- grep(group, rownames(env), value = T)
# 执行分组相关性分析
r_result <- corr.test(taxonomy[g, ], env[g, ], method = 'spearman')
# 提取r值
r <-
r_result$r %>%
melt() %>%
set_names(c('tax', 'Index', 'r'))
# 提取P值
p <-
r_result$p %>%
melt() %>%
set_names(c('tax', 'Index', 'P_value')) %>%
mutate(P_value_sig = case_when(P_value > 0.05 ~ " ",
P_value <= 0.05 & P_value > 0.01 ~ "*",
P_value <= 0.01 & P_value > 0.001 ~ "**",
P_value <= 0.001 ~ "***",
TRUE ~ NA_character_))
# 合并数据
data <- cbind(r,p) %>%
select(-(4:5)) %>%
mutate(group)
data
}
cal('A') %>% head()
cal('B') %>% head()
cal('C') %>% head()
Step4:相关性热图(“*”标记显著性)
# 数据合并
data <- rbind(cal('A'), cal('B'), cal('C'))
# 指定封面顺序
data$group <- factor(data$group, levels = c('A', 'B', 'C'), ordered=TRUE)
# 出图
ggplot(data,aes(x=Index, y=tax))+
geom_tile(aes(fill=r), color = 'white',alpha = 0.6) +
geom_text(aes(label = P_value_sig), color = 'black', size = 2) +
ggsci::scale_fill_gsea() +
theme(legend.position="top") +
theme +
facet_grid( ~ group, scales = 'free') +
labs(y = 'Taxonomy', x = 'Environment', title = 'Spearman Correlation Plot', size = '|rho|', color = 'rho')
ggsave('heatmap_corr1.png',height = 2, width = 6)
Step5:相关性热图(P值和“*”同时标记显著性)
# 数据整理
data <- rbind(cal('A'), cal('B'), cal('C')) %>%
mutate(P_value_sig2 = paste0(round(P_value, 2), ' ', P_value_sig))
theme_heatmap <-
theme_bw(base_size = 7) +
theme(panel.grid.major = element_blank(),
panel.grid.minor = element_blank(),
legend.key.size = unit(5, "pt"),
panel.border = element_blank(),
axis.ticks.x = element_blank(),
axis.ticks.y = element_blank(),
text = element_text(face='bold'),
axis.text.x = element_text(angle=45,vjust=1, hjust=1))
ggplot(data,aes(x=Index, y=tax)) +
geom_tile(aes(fill=r), alpha = 0.6) +
geom_text(aes(label = P_value_sig2), color = 'black', size = 1) +
ggsci::scale_fill_gsea() +
theme_heatmap +
coord_flip() +
facet_grid( ~ group, scale = 'free') +
labs(y = 'Taxonomy', x = 'Environment', title = 'Spearman Correlation Plot', size = '|rho|', color = 'rho')
ggsave('heatmap_corr2.png',height = 2.5, width = 6)
最后,我们可以通过绘制精美的气泡图来展示相关性分析结果:
Step6:气泡图展示相关性分析结果
ggplot(data,aes(x=Index, y=tax))+
geom_point(aes(color = r, size = abs(r))) +
geom_text(aes(label = P_value_sig2), color = 'black', size = 1) +
scale_size_area(max_size = 5) +
ggsci::scale_color_material('teal') +
theme_heatmap +
theme(legend.key.size = unit(6, "pt"),
strip.background = element_rect(fill = '#99CCCC')) +
facet_wrap( ~ group,strip.position = 'bottom') +
labs(y = 'Taxonomy', x = 'Environment', title = 'Spearman Correlation Plot', size = '|rho|', color = 'rho')
ggsave('heatmap_corr3.png',height = 2, width = 6)

关键词“相关性分析图”获取代码和数据
更多推荐
 已为社区贡献2条内容
已为社区贡献2条内容







所有评论(0)